ヒト幹細胞分化に関係する転写因子の網羅的な同定とその解析
AI を駆使した自在に細胞運命を制御する技術の開発に期待
2020年7月17日
慶應義塾大学医学部
国立成育医療研究センター
かずさDNA研究所
株式会社テンクー
株式会社DNAチップ研究所
報道関係者各位
このたび、慶應義塾大学医学部坂口光洋記念講座(システム医学)(教授:洪 実)、および国立成育医療研究センター(理事長:五十嵐 隆)、かずさDNA研究所(理事長:大石 道夫)、株式会社テンクー(代表取締役社長:西村 邦裕)、株式会社DNAチップ研究所(代表取締役社長:的場 亮)らの研究グループは、ヒト胚性幹(Embryonic Stem:ES)細胞を用いて、714遺伝子を個別に誘導しつつ、発現解析を行うことにより、幹細胞の分化に関係する転写因子(transcription factor:TF)を網羅的に同定することに成功しました。
得られた成果は、公的データベースと慶應義塾大学医学部坂口光洋記念講座ウェブサイトで公開されるとともに(本稿項目3参照)、樹立された遺伝子誘導できるヒトES細胞も、広く研究者に提供されます。これらのデータリソース・材料リソースは、ヒト遺伝子の機能を体系的に解析したもののなかでは、これまでになく大規模なもので、幹細胞研究のみならず、TFの機能解明に大きく貢献します。
本研究で得られた生物ビッグデータは、転写因子を同一プラットフォームで誘導したというユニークな特徴を持ち、人工知能(Artificial Intelligence:AI)などの数理的な解析に適しています。本データを用いることにより複雑なTF同士のネットワーク構造の解明が大きく進み、iPS細胞をはじめとしたヒト幹細胞から、望む細胞系譜を自在に生み出す技術開発につながることが期待されます。
本研究成果は、2020年5月19日付(英国時間)に、「Cell Reports」誌のオンライン版に掲載されました。
1.研究の背景と概要
ES(注1)細胞は、胚盤胞に移植することであらゆる臓器に分化する細胞で、人工多能性幹(induced pluripotent:iPS)細胞とともに、多能性幹細胞として知られています。近年、ヒト多能性幹細胞から、試験管内でさまざまな組織を作出し幅広い移植治療に使用する再生医療に期待がされています。一部の細胞・組織はすでに臨床研究が開始されていますが、多能性幹細胞から目的とする細胞への分化を制御する決定的な因子が不明なことが多いため、多能性幹細胞は一部の細胞系譜(注2)に応用されるにとどまり、大部分の細胞系譜については、まだ分化誘導法が確立していません。
転写因子(transcription factor:TF、注3)は、細胞運命(注4)に決定的な役割を果たすことが知られます。マウスなどの先行研究では、大規模なTF誘導実験/解析が行われていますが、ヒトにおいては体系的な大規模研究は行われておらず、TFと細胞運命の因果関係の多くが不明なままでした。
そこで本研究グループは、ヒトゲノムに存在する20~30%相当のTFを収集し、これらを主とする714遺伝子を対象に、ES細胞の細胞運命の決定における機能を推定する大規模な解析を行いました。薬剤により発現を制御できる遺伝子を組み込んだヒトES細胞を、714遺伝子について、それぞれ3つのクローンを作製し、合計2,135のヒトES細胞株を樹立しました。これを用いて、各導入遺伝子を発現させた際のヒトES細胞の形態変化を記録した26,998の顕微鏡画像を得ました(図1左下)。また、511遺伝子については、導入48時間後の遺伝子発現(トランスクリープトーム:注5)データを取得しました(図1右下)。
また、各TFの発現を誘導した後の細胞内トランスクリプトームを、公共データベースから取得したヒトのさまざまな臓器の細胞におけるトランスクリプトームと比較しスコア化したところ、特定の臓器の遺伝子発現の状態と類似性の高い状態を作り出すTFが同定されました(図2)。本研究によって、TFの機能が網羅的に解析され、ヒトの各臓器へ寄与するTFが一括して示されました。今後、各TFの機能を詳細に解析することで、細胞運命を決定づける分子機構が解明されると期待できます。
今回の研究では、1: 714遺伝子を薬剤で誘導できるヒトES細胞株(合計2,135株)、2: それらの形態変化を記録した顕微鏡データ(26,998ファイル)、3: RNAシーケンスおよびマイクロアレイによる511遺伝子を誘導した48時間後の発現データ(2,174データセット)が取得されました。取得された細胞株およびデータセットは、公的機関および研究室のウェブサイトで公開され(本稿項目3参照)、広く研究者コミュニティで活用されることが期待されます。これらのデータリソース・材料リソースは、ヒト遺伝子の機能を体系的に解析したもののなかでは、これまでになく大規模なもので、幹細胞研究のみならず、TFの機能解明に大きく貢献します。本研究で得られた生物ビッグデータは、転写因子を同一プラットフォームで誘導したユニークな特徴を持ち、人工知能(Artificial Intelligence:AI)などの数理的な解析に適しています。これらのデータの解析により、今後、複雑なTF同士のネットワーク構造の解明が大きく進み、iPS細胞をはじめとしたヒト幹細胞から、望む細胞系譜を自在に生み出す技術開発につながることが期待されます。
3.データおよび解析ツールの公開
本研究で得られた成果は以下で公開されています。
・マイクロアレイ解析(Study: JGAS00000000121)
Genotype-phenotype Archives:https://www.ddbj.nig.ac.jp/jga/index-e.html
・配列情報(DRA006296)
DDBJ Sequence Read Archive:https://www.ddbj.nig.ac.jp/dra/index-e.html
・顕微鏡画像
慶應義塾大学医学部坂口光洋記念講座Webサイト
本解析結果DB:http://www.systemsmedicine.jp/crest/plasmid/
また本研究で発現解析に使用したツールは、以下で公開されています。
・遺伝子発現解析ツール
ExAtlas:
https://lgsun.irp.nia.nih.gov/exatlas/
http://webtools.systemsmedicine.jp/
4.特記事項
本研究は、主として国立研究開発法人科学技術振興機構(JST)の戦略的創造研究推進事業(チーム型研究(CREST)『生命動態の理解と制御のための基盤技術の創出』研究領域)、国立研究開発法人日本医療研究開発機構(AMED)の再生医療実現拠点ネットワークプログラム(技術開発個別課題)「多能性幹細胞から多種類の分化細胞を、最短時間、高効率、高品質、大量、自在に生産するための基盤技術開発と産業化応用」、「慶應義塾坂口光洋記念システム医学講座」の支援により行われました。また、本研究は発現解析について、米国National Institute on Aging, National Institute of Healthの協力を得て行われました。
本研究成果に関連して、慶應義塾大学は「多能性幹細胞を所望の細胞型へ分化する方法」として特許出願を行っています。
5.論文
タイトル:Generation and profiling of 2,135 human ESC lines for the systematic analyses of cell states perturbed by inducing single transcription factors
日本語訳:単一転写因子による遺伝子摂動解析のための2135株のヒトトランスジェニックES細胞の樹立とその発現プロファイリング
著者名:中武悠樹、洪繁、Alexei Sharov、若林俊一、村上都湖、迫田実希、近澤奈々、大倉千明、佐藤紗恵子、伊東紀子、平山円、Siu San Mak、Lars Martin Jakt、上野友生、平塚健、松下美紗子、Sravan Kumar Goparaju、秋山智彦、石黒敬一郎、小田真由美、合田徳夫、梅澤明弘、阿久津英憲、西村邦裕、的場亮、小原收、洪実
掲載誌:「Cell Reports」オンライン版
DOI:10.1016/j.celrep.2020.107655
【用語解説】
(注1)ヒト胚性幹(Embryonic Stem:ES)細胞:発生初期の胚盤胞から樹立された培養細胞。人工多能性幹細胞(iPS細胞)と同様に、転写因子の誘導によって試験管の中でさまざまな細胞に分化可能である。樹立されている多能性幹細胞としては、ES細胞がより標準的とされている。
(注2)細胞系譜:一個の受精卵が分裂して成体になるまでの細胞の系統。おおまかには、外胚葉、中胚葉、内胚葉に分類され、さらに細分化される。
(注3)転写因子(Transcriptional Factor:TF):細胞核の中で行われる転写(RNAの発現)をつかさどるタンパク質。主にDNAに直接結合するものを指すが、報告によって定義が異なる。本研究では2018年のCell誌の総説をもとに転写因子を定義している。
(注4)細胞運命:ゲノムにコードされている情報に基づき、未分化な細胞が分化していく過程のこと。受精卵、胚盤胞に存在する幹細胞、体性幹細胞、の順に、細胞が分化できる範囲は限定されていき、成熟した細胞になると、細胞の状態が変化しにくくなり、細胞の機能が定まる。
(注5)トランスクリプトーム:細胞中に存在する全てのmRNAの総体を指す。これを対象とした網羅的な解析法として、マイクロアレイ解析、シーケンス解析が共に汎用されている。マイクロアレイは、小さな基盤に特定のDNA断片を数万箇所に固定し、これに吸着する核酸を測定することにより、膨大な種類の遺伝子の発現量を一度に解析する。シーケンス解析は、次世代シーケンスとも呼ばれ、サンプル中の核酸断片をPCRで増幅し、測定する。
(注6)ヘテロクロマチン:特定のゲノム領域に観察される凝縮されたクロマチン状態を指す。主に短い繰り返し配列から構成され、転写されにくいことが知られる。
※ご取材の際には、事前に下記までご一報くださいますようお願い申し上げます。
※本リリースは文部科学記者会、科学記者会、厚生労働記者会、厚生日比谷クラブ、千葉県政記者会、千葉民間放送テレビ記者クラブ、木更津記者クラブ、各社科学部等に送信しております。
【本発表資料のお問い合わせ先】
慶應義塾大学医学部 坂口光洋記念講座(システム医学) 教授 洪 実(こう みのる)
TEL:03-5843-6174 FAX:03-5843-6177
【AMED事業に関するお問い合わせ先】
国立研究開発法人 日本医療研究開発機構
再生・細胞医療・遺伝子治療事業部 再生医療研究開発課
TEL:03-6870-2220 E-mail:saisei“AT”amed.go.jp
【本リリースの発信元】
慶應義塾大学信濃町キャンパス総務課:鈴木・山崎
〒160-8582 東京都新宿区信濃町35
TEL:03-5363-3611 FAX:03-5363-3612
E-mail : med-koho@adst.keio.ac.jp Web : http://www.med.keio.ac.jp/
国立成育医療研究センター 企画戦略局 広報企画室:村上・近藤
TEL:03-3416-0181(内線 7783・7249) E-mail : koho@ncchd.go.jp
かずさDNA研究所 広報・研究推進グループ
〒292-0818 千葉県木更津市かずさ鎌足2-6-7
TEL:0438-52-3930 FAX:0438-52-3931
E-mail : kdri-kouhou@kazusa.or.jp Web : https://www.kazusa.or.jp
株式会社テンクー 担当:鈴木
〒113-0033東京都文京区本郷4−2−5
TEL: 03-3868-2374 E-mail: pr@xcoo.jp Web: https://xcoo.co.jp
株式会社DNAチップ研究所 総務部:大塚
〒105-0022東京都港区海岸1-15-1
スズエベイディアム 5階
TEL:03-5777-1700 FAX:03-5777-1702
E-mail: info@dna-chip.co.jp Web: http://www.dna-chip.co.jp/
※本リリースのカラー版をご希望の方は上記【本リリースの発信元】までご連絡ください。
慶應義塾大学医学部
国立成育医療研究センター
かずさDNA研究所
株式会社テンクー
株式会社DNAチップ研究所
報道関係者各位
このたび、慶應義塾大学医学部坂口光洋記念講座(システム医学)(教授:洪 実)、および国立成育医療研究センター(理事長:五十嵐 隆)、かずさDNA研究所(理事長:大石 道夫)、株式会社テンクー(代表取締役社長:西村 邦裕)、株式会社DNAチップ研究所(代表取締役社長:的場 亮)らの研究グループは、ヒト胚性幹(Embryonic Stem:ES)細胞を用いて、714遺伝子を個別に誘導しつつ、発現解析を行うことにより、幹細胞の分化に関係する転写因子(transcription factor:TF)を網羅的に同定することに成功しました。
得られた成果は、公的データベースと慶應義塾大学医学部坂口光洋記念講座ウェブサイトで公開されるとともに(本稿項目3参照)、樹立された遺伝子誘導できるヒトES細胞も、広く研究者に提供されます。これらのデータリソース・材料リソースは、ヒト遺伝子の機能を体系的に解析したもののなかでは、これまでになく大規模なもので、幹細胞研究のみならず、TFの機能解明に大きく貢献します。
本研究で得られた生物ビッグデータは、転写因子を同一プラットフォームで誘導したというユニークな特徴を持ち、人工知能(Artificial Intelligence:AI)などの数理的な解析に適しています。本データを用いることにより複雑なTF同士のネットワーク構造の解明が大きく進み、iPS細胞をはじめとしたヒト幹細胞から、望む細胞系譜を自在に生み出す技術開発につながることが期待されます。
本研究成果は、2020年5月19日付(英国時間)に、「Cell Reports」誌のオンライン版に掲載されました。
1.研究の背景と概要
ES(注1)細胞は、胚盤胞に移植することであらゆる臓器に分化する細胞で、人工多能性幹(induced pluripotent:iPS)細胞とともに、多能性幹細胞として知られています。近年、ヒト多能性幹細胞から、試験管内でさまざまな組織を作出し幅広い移植治療に使用する再生医療に期待がされています。一部の細胞・組織はすでに臨床研究が開始されていますが、多能性幹細胞から目的とする細胞への分化を制御する決定的な因子が不明なことが多いため、多能性幹細胞は一部の細胞系譜(注2)に応用されるにとどまり、大部分の細胞系譜については、まだ分化誘導法が確立していません。
転写因子(transcription factor:TF、注3)は、細胞運命(注4)に決定的な役割を果たすことが知られます。マウスなどの先行研究では、大規模なTF誘導実験/解析が行われていますが、ヒトにおいては体系的な大規模研究は行われておらず、TFと細胞運命の因果関係の多くが不明なままでした。
そこで本研究グループは、ヒトゲノムに存在する20~30%相当のTFを収集し、これらを主とする714遺伝子を対象に、ES細胞の細胞運命の決定における機能を推定する大規模な解析を行いました。薬剤により発現を制御できる遺伝子を組み込んだヒトES細胞を、714遺伝子について、それぞれ3つのクローンを作製し、合計2,135のヒトES細胞株を樹立しました。これを用いて、各導入遺伝子を発現させた際のヒトES細胞の形態変化を記録した26,998の顕微鏡画像を得ました(図1左下)。また、511遺伝子については、導入48時間後の遺伝子発現(トランスクリープトーム:注5)データを取得しました(図1右下)。
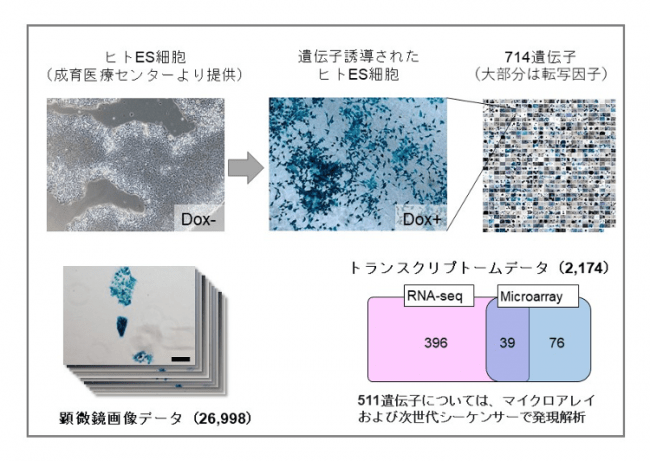 【図1】新たに確立されたバイオリソースおよびデータリソース
【図1】新たに確立されたバイオリソースおよびデータリソース
また、各TFの発現を誘導した後の細胞内トランスクリプトームを、公共データベースから取得したヒトのさまざまな臓器の細胞におけるトランスクリプトームと比較しスコア化したところ、特定の臓器の遺伝子発現の状態と類似性の高い状態を作り出すTFが同定されました(図2)。本研究によって、TFの機能が網羅的に解析され、ヒトの各臓器へ寄与するTFが一括して示されました。今後、各TFの機能を詳細に解析することで、細胞運命を決定づける分子機構が解明されると期待できます。
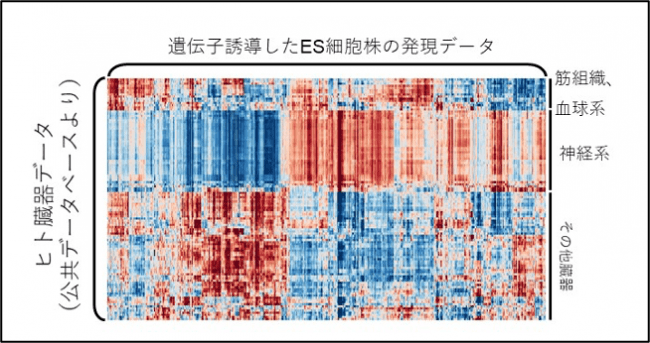 【図2】遺伝子誘導したES細胞株とヒト組織の相関を算出し、可視化
【図2】遺伝子誘導したES細胞株とヒト組織の相関を算出し、可視化
今回の研究では、1: 714遺伝子を薬剤で誘導できるヒトES細胞株(合計2,135株)、2: それらの形態変化を記録した顕微鏡データ(26,998ファイル)、3: RNAシーケンスおよびマイクロアレイによる511遺伝子を誘導した48時間後の発現データ(2,174データセット)が取得されました。取得された細胞株およびデータセットは、公的機関および研究室のウェブサイトで公開され(本稿項目3参照)、広く研究者コミュニティで活用されることが期待されます。これらのデータリソース・材料リソースは、ヒト遺伝子の機能を体系的に解析したもののなかでは、これまでになく大規模なもので、幹細胞研究のみならず、TFの機能解明に大きく貢献します。本研究で得られた生物ビッグデータは、転写因子を同一プラットフォームで誘導したユニークな特徴を持ち、人工知能(Artificial Intelligence:AI)などの数理的な解析に適しています。これらのデータの解析により、今後、複雑なTF同士のネットワーク構造の解明が大きく進み、iPS細胞をはじめとしたヒト幹細胞から、望む細胞系譜を自在に生み出す技術開発につながることが期待されます。
3.データおよび解析ツールの公開
本研究で得られた成果は以下で公開されています。
・マイクロアレイ解析(Study: JGAS00000000121)
Genotype-phenotype Archives:https://www.ddbj.nig.ac.jp/jga/index-e.html
・配列情報(DRA006296)
DDBJ Sequence Read Archive:https://www.ddbj.nig.ac.jp/dra/index-e.html
・顕微鏡画像
慶應義塾大学医学部坂口光洋記念講座Webサイト
本解析結果DB:http://www.systemsmedicine.jp/crest/plasmid/
また本研究で発現解析に使用したツールは、以下で公開されています。
・遺伝子発現解析ツール
ExAtlas:
https://lgsun.irp.nia.nih.gov/exatlas/
http://webtools.systemsmedicine.jp/
4.特記事項
本研究は、主として国立研究開発法人科学技術振興機構(JST)の戦略的創造研究推進事業(チーム型研究(CREST)『生命動態の理解と制御のための基盤技術の創出』研究領域)、国立研究開発法人日本医療研究開発機構(AMED)の再生医療実現拠点ネットワークプログラム(技術開発個別課題)「多能性幹細胞から多種類の分化細胞を、最短時間、高効率、高品質、大量、自在に生産するための基盤技術開発と産業化応用」、「慶應義塾坂口光洋記念システム医学講座」の支援により行われました。また、本研究は発現解析について、米国National Institute on Aging, National Institute of Healthの協力を得て行われました。
本研究成果に関連して、慶應義塾大学は「多能性幹細胞を所望の細胞型へ分化する方法」として特許出願を行っています。
5.論文
タイトル:Generation and profiling of 2,135 human ESC lines for the systematic analyses of cell states perturbed by inducing single transcription factors
日本語訳:単一転写因子による遺伝子摂動解析のための2135株のヒトトランスジェニックES細胞の樹立とその発現プロファイリング
著者名:中武悠樹、洪繁、Alexei Sharov、若林俊一、村上都湖、迫田実希、近澤奈々、大倉千明、佐藤紗恵子、伊東紀子、平山円、Siu San Mak、Lars Martin Jakt、上野友生、平塚健、松下美紗子、Sravan Kumar Goparaju、秋山智彦、石黒敬一郎、小田真由美、合田徳夫、梅澤明弘、阿久津英憲、西村邦裕、的場亮、小原收、洪実
掲載誌:「Cell Reports」オンライン版
DOI:10.1016/j.celrep.2020.107655
【用語解説】
(注1)ヒト胚性幹(Embryonic Stem:ES)細胞:発生初期の胚盤胞から樹立された培養細胞。人工多能性幹細胞(iPS細胞)と同様に、転写因子の誘導によって試験管の中でさまざまな細胞に分化可能である。樹立されている多能性幹細胞としては、ES細胞がより標準的とされている。
(注2)細胞系譜:一個の受精卵が分裂して成体になるまでの細胞の系統。おおまかには、外胚葉、中胚葉、内胚葉に分類され、さらに細分化される。
(注3)転写因子(Transcriptional Factor:TF):細胞核の中で行われる転写(RNAの発現)をつかさどるタンパク質。主にDNAに直接結合するものを指すが、報告によって定義が異なる。本研究では2018年のCell誌の総説をもとに転写因子を定義している。
(注4)細胞運命:ゲノムにコードされている情報に基づき、未分化な細胞が分化していく過程のこと。受精卵、胚盤胞に存在する幹細胞、体性幹細胞、の順に、細胞が分化できる範囲は限定されていき、成熟した細胞になると、細胞の状態が変化しにくくなり、細胞の機能が定まる。
(注5)トランスクリプトーム:細胞中に存在する全てのmRNAの総体を指す。これを対象とした網羅的な解析法として、マイクロアレイ解析、シーケンス解析が共に汎用されている。マイクロアレイは、小さな基盤に特定のDNA断片を数万箇所に固定し、これに吸着する核酸を測定することにより、膨大な種類の遺伝子の発現量を一度に解析する。シーケンス解析は、次世代シーケンスとも呼ばれ、サンプル中の核酸断片をPCRで増幅し、測定する。
(注6)ヘテロクロマチン:特定のゲノム領域に観察される凝縮されたクロマチン状態を指す。主に短い繰り返し配列から構成され、転写されにくいことが知られる。
※ご取材の際には、事前に下記までご一報くださいますようお願い申し上げます。
※本リリースは文部科学記者会、科学記者会、厚生労働記者会、厚生日比谷クラブ、千葉県政記者会、千葉民間放送テレビ記者クラブ、木更津記者クラブ、各社科学部等に送信しております。
【本発表資料のお問い合わせ先】
慶應義塾大学医学部 坂口光洋記念講座(システム医学) 教授 洪 実(こう みのる)
TEL:03-5843-6174 FAX:03-5843-6177
【AMED事業に関するお問い合わせ先】
国立研究開発法人 日本医療研究開発機構
再生・細胞医療・遺伝子治療事業部 再生医療研究開発課
TEL:03-6870-2220 E-mail:saisei“AT”amed.go.jp
【本リリースの発信元】
慶應義塾大学信濃町キャンパス総務課:鈴木・山崎
〒160-8582 東京都新宿区信濃町35
TEL:03-5363-3611 FAX:03-5363-3612
E-mail : med-koho@adst.keio.ac.jp Web : http://www.med.keio.ac.jp/
国立成育医療研究センター 企画戦略局 広報企画室:村上・近藤
TEL:03-3416-0181(内線 7783・7249) E-mail : koho@ncchd.go.jp
かずさDNA研究所 広報・研究推進グループ
〒292-0818 千葉県木更津市かずさ鎌足2-6-7
TEL:0438-52-3930 FAX:0438-52-3931
E-mail : kdri-kouhou@kazusa.or.jp Web : https://www.kazusa.or.jp
株式会社テンクー 担当:鈴木
〒113-0033東京都文京区本郷4−2−5
TEL: 03-3868-2374 E-mail: pr@xcoo.jp Web: https://xcoo.co.jp
株式会社DNAチップ研究所 総務部:大塚
〒105-0022東京都港区海岸1-15-1
スズエベイディアム 5階
TEL:03-5777-1700 FAX:03-5777-1702
E-mail: info@dna-chip.co.jp Web: http://www.dna-chip.co.jp/
※本リリースのカラー版をご希望の方は上記【本リリースの発信元】までご連絡ください。
このプレスリリースには、メディア関係者向けの情報があります
メディアユーザーログイン既に登録済みの方はこちら
メディアユーザー登録を行うと、企業担当者の連絡先や、イベント・記者会見の情報など様々な特記情報を閲覧できます。※内容はプレスリリースにより異なります。
すべての画像
- 種類
- その他
- 関連リンク
- https://xcoo.co.jp
- ダウンロード