ナノサイズの共進化:反復DNA配列と転写酵素 -熱揺らぎを利用する酵素機構の解明に貢献-
千葉大学真菌医学研究センター今清水正彦元特任助教(前、米国NIH、現、東京大学医科学研究所)、高橋弘喜准教授、イスラエル国ネゲヴ・ベン=グリオン大学David B. Lukatsky准教授らの共同研究グループは、熱揺らぎを、ゲノムにコードされた反復性と組み合わせて利用する転写酵素の機構を発見することに成功しました。成果は、米国の科学雑誌『Proceedings of the National Academy of Sciences』に掲載されるのに先立ち、オンライン版(11月8日付け、日本時間)に掲載されます。
①RNAPによる転写伸長反応の調節機構は未だ不明点が多い
RNAポリメラーゼ(RNAP)は、ゲノムDNAの塩基情報をRNAに転写する酵素です。RNAPによる転写伸長反応の基本的な調節機構は、DNA配列依存的な「一時停止」ですが、その仕組みは良く理解されていません。この配列依存的な一時停止は、遺伝子発現の多岐にわたる調節機能の根幹を担う一方、停止が長引いた場合は疾患や加齢に繋がることも知られています[1]。
生物ナノマシンであるRNAPは熱揺らぎを必ず持ち、反応に熱運動は必須です[2]。配列依存的な一時停止反応にも、どこかの過程で熱揺らぎを利用すると考えられます。しかし、ランダムな熱揺らぎが、ゲノム全体で統制のとれた転写調節機構にどのように利用されるのでしょうか?
②反復DNA配列が謎を解く鍵
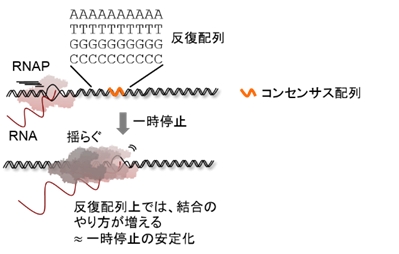
共同研究グループは、塩基の種類に依らない配列反復性の指標[3]と、実験的に検出された転写一時停止の強さに相関があることを見出しました。高い反復性のある配列上では、熱揺らぎにより変換される弱い結合のやり方の総数が増えるため、そのような配列上では、弱い結合の足し合わせの結果、強い転写の一時停止が観察されると考えられます[4]。同グループは、この考えの正当性を生化学的に立証し、生物進化の過程でゲノムDNAに埋め込まれた反復配列要素が、揺らぎ効果に配列依存的な「偏り」を造り出し、それがアンサンブル平均とした時の転写調節機構になったと結論づけました。
③今後の期待
本研究で得られた基礎概念は、基礎科学に留まらず、核酸創薬、バイオセンサーといったナノバイオテクノロジー技術の基盤となる核酸—標的分子の結合親和性をデザインするための方法に繋がることが期待できます。
遺伝子発現はDNAからRNAへの転写から始まり、その反応はRNAPによって触媒されます。RNAPと2本鎖DNA、RNA-DNAハイブリッドから構成される転写複合体は、分子機構の解明が最も進んだ生物ナノマシンの1つです[2]。高い正確性を維持した転写伸長反応の機構モデルとして、正しい基質のRNAP活性中心への結合と、RNAPの1方向性揺らぎ運動のカップリングが提案されてきました(Bar-Nahum et al., Cell 2005, 120:183–193)。RNAPは、DNA上のランダムな揺らぎ運動で前にも後ろにも進みますが、鋳型DNA情報に即した基質の結合が、後ろ向き運動を阻害するため、前向き運動と同時に読み誤りを防ぐことができます。
転写反応におけるRNAPは、電車が均質な単位から成るレール上を走るのとは異なり、不均質なATGCの組み合わせからなるDNA配列上を走ります。その結果、RNAPの配列依存的な一時停止が起こり、これが転写の基本調節機構になります(Imashimizu et al., Transcription 2014, 5:e28285)。この配列依存的な一時停止を加味した形で、上記の揺らぎ運動を用いた転写機構は説明されていませんでした。
本研究グループは、これまでに大腸菌細胞において転写の一時停止を起こすコンセンサス配列(pause-inducing element (PIE))を同定しました(Imashimizu et al., Genome Biol 2015, 16:98)。PIEの塩基情報の重み行列を使い、大腸菌ゲノムでPIEと同等以上の情報量をもつが一時停止が起こらない配列(false PIE)を調べた所、RNAPが一時停止したPIEは全転写領域のわずか3%であり、残りの97%はfalse PIEでした。これはRNAPの一時停止はPIEの塩基配列情報だけでは決まらず、未知の調節因子に強く支配されていることを意味します。本研究では、その因子を反復配列要素として同定しました。そして、揺らぎ運動を利用する転写伸長と一時停止機構の一般的な説明が可能になりました。
本研究では、ハイスループットシーケンシング法を駆使して、大腸菌細胞において様々な強さの転写の一時停止をゲノムワイドに検出する方法を確立しました(Imashimizu et al., Genome Biol 2015)。この方法では、沢山の転写複合体由来の分子情報を個々に扱えます。したがって、個々の複合体から成る確率分布を用いて、転写機構を統計的に解釈することができます。
本研究の最大の特徴は、上記の実験測定結果と統計力学モデル(Sela & Lukatsky, Biophysical J 2011, 101:160–166)を組み合わせ、転写調節の分子機構に熱揺らぎの影響を踏まえたことです。Lukatskyらの統計力学モデルでは、タンパク質—DNA結合を塩基毎の結合の揺らぎだけで定義しており、結合サイト周辺の塩基配列に反復性がある場合に、計算される平均自由エネルギーが小さくなります。これまでに、様々な転写因子の結合サイトを予測してきました(Afek et al., PNAS 2014, 111:17140–17145)。本研究では、この統計力学モデルによる予測の正当性を、in vitro精製再構成系[5]を構築して実験的に立証しました。
本研究独自の統計的アプローチにより、従来のコンセンサス配列という概念では予測し得なかった転写の一時停止の運命を予測できるようになりました。一方、この予測は、沢山の配列がある時の統計的な予測であって、必ずしも個々に全て正しいわけではありません。この考え方は、現在の生物学に十分に浸透していませんが、熱揺らぎを利用せざるを得ない生物のナノシステムの現実とは合っているため、今後の浸透が予想されます。
ナノサイズの核酸やタンパク質から成る複合体は、熱揺らぎの影響が大きく、水溶液中での構造は、水分子との衝突により不均一です。そのため、1機能が1構造に対応するという従来の構造生物学で良く用いられてきた前提は、正しくない場合があります。一方で、この構造(微視的状態)の不均一性は、核酸—タンパク質結合の親和性(あるいは核酸—タンパク質間の滑り運動)に一般的に寄与すると考えられます。転写について言えば、伸長反応に限らず、開始反応の一時停止も配列から予測することができると考えています。開始の分岐反応と関連して起こる一時停止のため、実際に機能する転写のプロモーター配列は、大腸菌ですら未だに十分に予測できていません。
この構造不均一性に基づく機能は、RNAータンパク質複合体でも有効と考えています。RNAは、分子内結合を急速に変えられるので高い構造不均一性を持つことは良く知られています。特に近年、ノンコーディング RNAとタンパク質から成る複合体の新しい機能が相次いで報告されており、その機能を配列から予測することにも役立つ可能性があります。
本研究で得られた基礎概念は、基礎科学に留まらず、核酸創薬、バイオセンサーといったナノバイオテクノロジー技術の基盤となる核酸—標的分子の結合親和性をデザインするための方法に繋がることが期待できます。
<タイトル>
Control of Transcriptional Pausing by Biased Thermal Fluctuations on Repetitive Genomic Sequences
<著者名>
Imashimizu, Masahiko; Afek, Ariel; Takahashi, Hiroki; Lubkowska, Lucyna; Lukatsky, David B.
<雑誌>
Proceedings of the National Academy of Sciences
<DOI>
10.1073/pnas.1607760113
[1]転写伸長の一時停止の生物学的意義
例えば、真核細胞のRNAPIIは転写を開始した直後に一時的に伸長反応を停止することにより、開いたクロマチン状態を維持し、環境シグナルに即応してRNA鎖を伸長することができます。一方で、長い伸長停止を起こしたRNAPは、DNA上で複製中のDNAポリメラーゼと衝突します。この結果できるRNA-DNAハイブリッドがDNA切断を誘起する場合があります。
[2]RNAPのナノバイオロジーについては、嶋本伸雄京都産業大学客員教授の総説(Shimamoto, Chem Rev 2013, 113:8400–8422)に詳しく書かれています。
[3]塩基の種類に依らない反復配列の例は、AAAAA…TTTTT…GGGGG…CCCCCのようなホモポリマーの繰り返し配列です。ここで用いた反復配列の指標は、Lukatskyらの非コンセンサス配列におけるタンパク質—DNA結合の自由エネルギーで定義されたものです(Afek et al., PNAS 2014, 111:17140–17145)。
[4]具体的に観察された現象は、一時停止したRNAP分子の上流DNAへの一次元拡散(diffusive backtracking)です。
[5]in vitro精製再構成実験は、生体反応の中から、目的の反応に関与する要素(本研究では大腸菌から精製したRNAPと合成DNA/RNA)だけを抽出・再構成した試験官内において、その反応機構を調べる方法です。
<研究内容に関するお問合せ>
東京大学医科学研究所 特任講師 今清水正彦(いましみず まさひこ)
TEL:03-5449-5307 FAX:03-5449-5415
E-mail:imashi@ims.u-tokyo.ac.jp
- 研究成果の概要
 熱揺らぎとゲノム反復配列を組み合わせた転写調節機構
熱揺らぎとゲノム反復配列を組み合わせた転写調節機構
①RNAPによる転写伸長反応の調節機構は未だ不明点が多い
RNAポリメラーゼ(RNAP)は、ゲノムDNAの塩基情報をRNAに転写する酵素です。RNAPによる転写伸長反応の基本的な調節機構は、DNA配列依存的な「一時停止」ですが、その仕組みは良く理解されていません。この配列依存的な一時停止は、遺伝子発現の多岐にわたる調節機能の根幹を担う一方、停止が長引いた場合は疾患や加齢に繋がることも知られています[1]。
生物ナノマシンであるRNAPは熱揺らぎを必ず持ち、反応に熱運動は必須です[2]。配列依存的な一時停止反応にも、どこかの過程で熱揺らぎを利用すると考えられます。しかし、ランダムな熱揺らぎが、ゲノム全体で統制のとれた転写調節機構にどのように利用されるのでしょうか?
②反復DNA配列が謎を解く鍵
共同研究グループは、塩基の種類に依らない配列反復性の指標[3]と、実験的に検出された転写一時停止の強さに相関があることを見出しました。高い反復性のある配列上では、熱揺らぎにより変換される弱い結合のやり方の総数が増えるため、そのような配列上では、弱い結合の足し合わせの結果、強い転写の一時停止が観察されると考えられます[4]。同グループは、この考えの正当性を生化学的に立証し、生物進化の過程でゲノムDNAに埋め込まれた反復配列要素が、揺らぎ効果に配列依存的な「偏り」を造り出し、それがアンサンブル平均とした時の転写調節機構になったと結論づけました。
③今後の期待
本研究で得られた基礎概念は、基礎科学に留まらず、核酸創薬、バイオセンサーといったナノバイオテクノロジー技術の基盤となる核酸—標的分子の結合親和性をデザインするための方法に繋がることが期待できます。
- 背景
遺伝子発現はDNAからRNAへの転写から始まり、その反応はRNAPによって触媒されます。RNAPと2本鎖DNA、RNA-DNAハイブリッドから構成される転写複合体は、分子機構の解明が最も進んだ生物ナノマシンの1つです[2]。高い正確性を維持した転写伸長反応の機構モデルとして、正しい基質のRNAP活性中心への結合と、RNAPの1方向性揺らぎ運動のカップリングが提案されてきました(Bar-Nahum et al., Cell 2005, 120:183–193)。RNAPは、DNA上のランダムな揺らぎ運動で前にも後ろにも進みますが、鋳型DNA情報に即した基質の結合が、後ろ向き運動を阻害するため、前向き運動と同時に読み誤りを防ぐことができます。
転写反応におけるRNAPは、電車が均質な単位から成るレール上を走るのとは異なり、不均質なATGCの組み合わせからなるDNA配列上を走ります。その結果、RNAPの配列依存的な一時停止が起こり、これが転写の基本調節機構になります(Imashimizu et al., Transcription 2014, 5:e28285)。この配列依存的な一時停止を加味した形で、上記の揺らぎ運動を用いた転写機構は説明されていませんでした。
本研究グループは、これまでに大腸菌細胞において転写の一時停止を起こすコンセンサス配列(pause-inducing element (PIE))を同定しました(Imashimizu et al., Genome Biol 2015, 16:98)。PIEの塩基情報の重み行列を使い、大腸菌ゲノムでPIEと同等以上の情報量をもつが一時停止が起こらない配列(false PIE)を調べた所、RNAPが一時停止したPIEは全転写領域のわずか3%であり、残りの97%はfalse PIEでした。これはRNAPの一時停止はPIEの塩基配列情報だけでは決まらず、未知の調節因子に強く支配されていることを意味します。本研究では、その因子を反復配列要素として同定しました。そして、揺らぎ運動を利用する転写伸長と一時停止機構の一般的な説明が可能になりました。
- 研究手法と成果
本研究では、ハイスループットシーケンシング法を駆使して、大腸菌細胞において様々な強さの転写の一時停止をゲノムワイドに検出する方法を確立しました(Imashimizu et al., Genome Biol 2015)。この方法では、沢山の転写複合体由来の分子情報を個々に扱えます。したがって、個々の複合体から成る確率分布を用いて、転写機構を統計的に解釈することができます。
本研究の最大の特徴は、上記の実験測定結果と統計力学モデル(Sela & Lukatsky, Biophysical J 2011, 101:160–166)を組み合わせ、転写調節の分子機構に熱揺らぎの影響を踏まえたことです。Lukatskyらの統計力学モデルでは、タンパク質—DNA結合を塩基毎の結合の揺らぎだけで定義しており、結合サイト周辺の塩基配列に反復性がある場合に、計算される平均自由エネルギーが小さくなります。これまでに、様々な転写因子の結合サイトを予測してきました(Afek et al., PNAS 2014, 111:17140–17145)。本研究では、この統計力学モデルによる予測の正当性を、in vitro精製再構成系[5]を構築して実験的に立証しました。
本研究独自の統計的アプローチにより、従来のコンセンサス配列という概念では予測し得なかった転写の一時停止の運命を予測できるようになりました。一方、この予測は、沢山の配列がある時の統計的な予測であって、必ずしも個々に全て正しいわけではありません。この考え方は、現在の生物学に十分に浸透していませんが、熱揺らぎを利用せざるを得ない生物のナノシステムの現実とは合っているため、今後の浸透が予想されます。
- 今後の期待
ナノサイズの核酸やタンパク質から成る複合体は、熱揺らぎの影響が大きく、水溶液中での構造は、水分子との衝突により不均一です。そのため、1機能が1構造に対応するという従来の構造生物学で良く用いられてきた前提は、正しくない場合があります。一方で、この構造(微視的状態)の不均一性は、核酸—タンパク質結合の親和性(あるいは核酸—タンパク質間の滑り運動)に一般的に寄与すると考えられます。転写について言えば、伸長反応に限らず、開始反応の一時停止も配列から予測することができると考えています。開始の分岐反応と関連して起こる一時停止のため、実際に機能する転写のプロモーター配列は、大腸菌ですら未だに十分に予測できていません。
この構造不均一性に基づく機能は、RNAータンパク質複合体でも有効と考えています。RNAは、分子内結合を急速に変えられるので高い構造不均一性を持つことは良く知られています。特に近年、ノンコーディング RNAとタンパク質から成る複合体の新しい機能が相次いで報告されており、その機能を配列から予測することにも役立つ可能性があります。
本研究で得られた基礎概念は、基礎科学に留まらず、核酸創薬、バイオセンサーといったナノバイオテクノロジー技術の基盤となる核酸—標的分子の結合親和性をデザインするための方法に繋がることが期待できます。
- 論文情報
<タイトル>
Control of Transcriptional Pausing by Biased Thermal Fluctuations on Repetitive Genomic Sequences
<著者名>
Imashimizu, Masahiko; Afek, Ariel; Takahashi, Hiroki; Lubkowska, Lucyna; Lukatsky, David B.
<雑誌>
Proceedings of the National Academy of Sciences
<DOI>
10.1073/pnas.1607760113
- 補足説明
[1]転写伸長の一時停止の生物学的意義
例えば、真核細胞のRNAPIIは転写を開始した直後に一時的に伸長反応を停止することにより、開いたクロマチン状態を維持し、環境シグナルに即応してRNA鎖を伸長することができます。一方で、長い伸長停止を起こしたRNAPは、DNA上で複製中のDNAポリメラーゼと衝突します。この結果できるRNA-DNAハイブリッドがDNA切断を誘起する場合があります。
[2]RNAPのナノバイオロジーについては、嶋本伸雄京都産業大学客員教授の総説(Shimamoto, Chem Rev 2013, 113:8400–8422)に詳しく書かれています。
[3]塩基の種類に依らない反復配列の例は、AAAAA…TTTTT…GGGGG…CCCCCのようなホモポリマーの繰り返し配列です。ここで用いた反復配列の指標は、Lukatskyらの非コンセンサス配列におけるタンパク質—DNA結合の自由エネルギーで定義されたものです(Afek et al., PNAS 2014, 111:17140–17145)。
[4]具体的に観察された現象は、一時停止したRNAP分子の上流DNAへの一次元拡散(diffusive backtracking)です。
[5]in vitro精製再構成実験は、生体反応の中から、目的の反応に関与する要素(本研究では大腸菌から精製したRNAPと合成DNA/RNA)だけを抽出・再構成した試験官内において、その反応機構を調べる方法です。
- お問合せ
<研究内容に関するお問合せ>
東京大学医科学研究所 特任講師 今清水正彦(いましみず まさひこ)
TEL:03-5449-5307 FAX:03-5449-5415
E-mail:imashi@ims.u-tokyo.ac.jp
 <機関窓口>
<機関窓口>
千葉大学 企画総務部渉外企画課広報室
TEL:043-290-2232 FAX:043-284-2550
E-mail:koho-hp@office.chiba-u.jp
すべての画像
