神経-肝型ミトコンドリアDNA枯渇症の新規原因遺伝子を特定
~ 世界初の症例を報告 ~
■ 本研究成果のポイント
神経-肝型ミトコンドリアDNA枯渇症の原因としてMICOS10 遺伝子を同定した
全ゲノム解析とRNAシークエンシングを用いた複合的解析によって原因の特定に至った
神経-肝型ミトコンドリアDNA枯渇症の原因としてMICOS複合体(*3)が関与していることが明らかとなった
■ 背景
ミトコンドリアの機能異常が原因となる病気を総称してミトコンドリア病と呼んでいます。この疾患の発症年齢や症状、遺伝形式は多岐に渡っており、臨床的また遺伝的に診断が非常に難しい疾患です。
岡﨑教授および村山教授らの研究グループは十数年にわたり、埼玉医科大学ゲノム医療科・小児科(大竹明 名誉教授)と共同で、ミトコンドリア病の生化学診断や遺伝子診断に取り組んできました。ミトコンドリアDNA枯渇症はミトコンドリア病の一つであり、ミトコンドリアDNA量が臓器で減少することを特徴とします。また、肝臓型や神経型、筋型、心筋型のミトコンドリアDNA枯渇症が報告されています。多くのミトコンドリアDNA枯渇症はミトコンドリアDNAの複製やミトコンドリアDNAの材料となるヌクレオチドの合成に関わるものでしたが、今回の報告では、MICOS複合体というミトコンドリアの内膜構造の形成に関わるMICOS10 遺伝子に異常があることを新たに見出しました。
■ 内容
今回、ミトコンドリア病の未解決症例を対象として、全ゲノム解析およびRNAシークエンスを行いました。対象となる症例は肝臓と神経に主な症状を呈し、肝臓においてミトコンドリアDNA量の低下が観察され、ミトコンドリアDNA枯渇症と診断されていました。従来のゲノム解析方法である全エクソーム解析を用いて原因探索を行いましたが、原因となる遺伝子異常を見つけることができませんでした。全ゲノム解析を行うことで、MICOS10 遺伝子にヘテロ接合のc.173G>C (p.Cys58Ser)の遺伝子異常を見出しました。AlphaFold2(*4)の予測によって、このアミノ酸残基は立体構造形成に重要であるジスルフィド結合を担うものであることがわかりました(図1)。また、エクソン1を巻き込む4.6k塩基対ほどの欠失を有することも明らかにしました(図2)。RNAシークエンスのデータから、 エクソン1を欠失している染色体からはMICOS10 遺伝子のmRNAが発現していないことを明らかにしました。つまり、c.173G>Cが存在する染色体からしか、MICOS10 遺伝子のmRNAが発現しておらず、そのmRNAの翻訳産物はp.Cys58Serの変化により、タンパク質機能が損なわれていると考えられました。
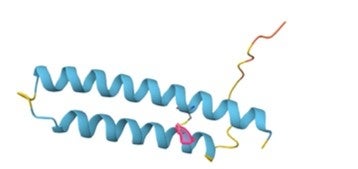
図1 MICOS10 遺伝子のバリアントとタンパク質構造
c.173G>Cの遺伝子異常は、タンパク質ではp.Cys58Serの異常となります。p.Cys58Ser は58番目のシステインがセリンに変化していることを表しています。Cys58(ピンク色)は図に示すように、向かい合うシステインとジスルフィド結合を形成する重要なアミノ酸残基となっていることをAlphaFold2のタンパク質立体構造予測が示しています。
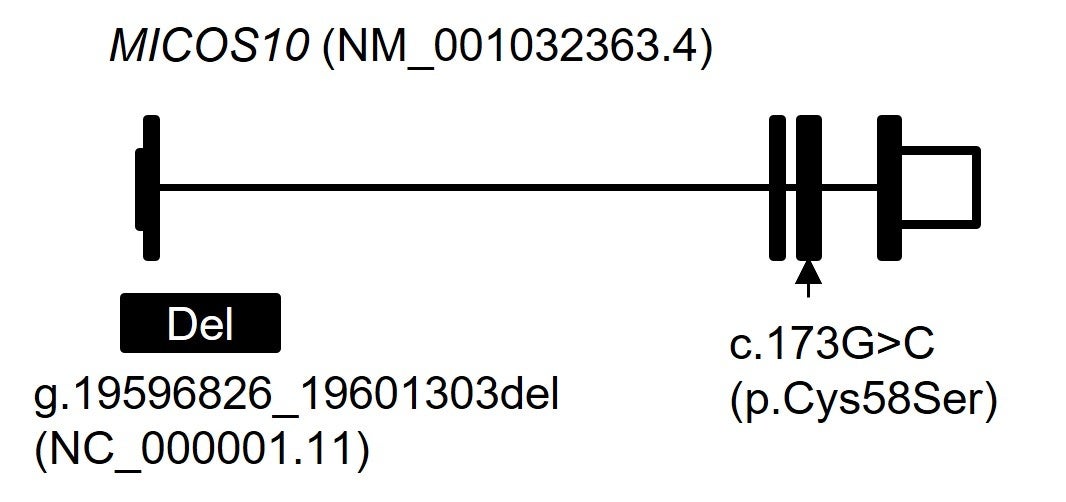
図2 MICOS10 遺伝子の遺伝子異常
全ゲノム解析の結果、 c.173G>C (p.Cys58Ser)に加えて、エクソン1を巻き込む4.6k塩基対の欠失(Del)を見出しました。横線はイントロン、黒塗りの四角はエクソン、白抜きの四角は非翻訳領域を示しています。
患者由来線維芽細胞において、MICOS10 遺伝子から発現するタンパク質であるMIC10の量が著しく減少していることも明らかにしました(図3)。図3に示すように、MIC10はMICOS複合体の一つのコンポーネントであり、この消失はミトコンドリア内膜に存在するクリステ構造に大きな影響を与えると考えられました。
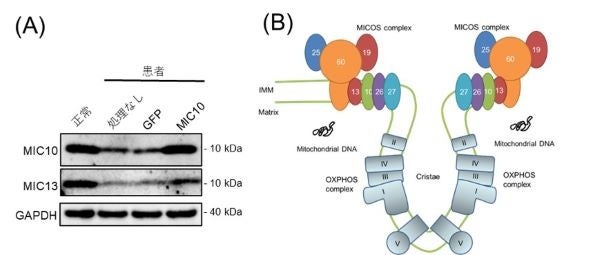
図3 MIC10タンパク質の消失およびMICOS複合体
(A)患者由来線維芽細胞では、MIC10タンパク質およびMIC13タンパク質が減少していました。それは、MICOS10 遺伝子を細胞内に戻すことで回復されました。
(B)MICOS複合体は、ミトコンドリア内膜(IMM)に存在し、MIC10,13,19,25,26,27,60から構成されています。これらは、クリステ構造を形作るのに重要な役割を担っています。
電子顕微鏡により、ミトコンドリアクリステの形態変化を観察しました。その結果、患者由来線維芽細胞において、クリステが消失しているミトコンドリアが多数観察されました(図4)。さらに、正常型のMICOS10を患者由来線維芽細胞に発現させることによって、クリステ形態の異常を回復できることも明らかにしました。また、それに伴ってミトコンドリアにおける酸素消費量も回復したことが確認されました。
以上より、神経-肝型ミトコンドリアDNA枯渇症の新規の原因遺伝としてMICOS10 遺伝子異常を特定することができました。
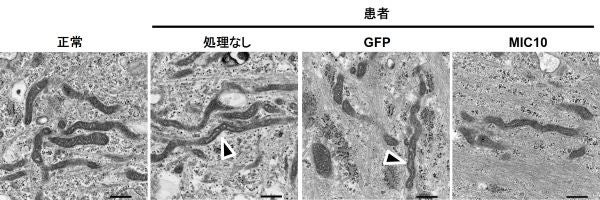
図4 クリステ形成の異常
患者由来線維芽細胞を電子顕微鏡により観察することで、クリステ形成の異常を観察しました(矢印)。また、MIC10を細胞に戻すことで、この異常は回復されました。
■ 今後の展開
本成果において、神経-肝型ミトコンドリアDNA枯渇症の原因として、MICOS10 遺伝子の異常を世界で初めて報告しました。また、先行研究では、MICOS13 遺伝子を神経-肝型ミトコンドリアDNA枯渇症の原因として報告していました(参考文献)。MICOS13 遺伝子はMIC13タンパク質をコードしており(図3)、MICOS複合体とミトコンドリアDNA枯渇症の関連が示唆されていました。今回の発見においても、MICOS複合体のコンポーネントであるMIC10との関連が示され、MICOS複合体とミトコンドリアDNA枯渇症の関連が深いことが明らかになりました。これらは、ミトコンドリアクリステ形成が、ミトコンドリアDNAの維持に重要であることを示しています。現在、新規治療薬として期待されているMA-5は、ミトコンドリアクリステ形成を促進するものであり、これがミトコンドリアDNA枯渇に対しても効果を発揮する可能性を本研究が示唆しています。
■ 用語解説
*1 全ゲノム解析
全ゲノム解析(Whole genome sequencing; WGS)は、文字通り全ゲノムを対象とした、配列を解読する解析手法です。全エクソーム解析で対象としない遺伝子間領域やイントロンまでも解析範囲となるため、全エクソーム解析では分からない遺伝子の異常を明らかにすることができます。
*2 RNAシークエンス
オミクス解析のひとつであるトランスクリプトミクスはRNAの発現を対象とした解析です。トランスクリプトミクスの解析方法として、RNAシーケンスが最も主流な方法となっています。主にメッセンジャーRNA(mRNA)を対象として、全遺伝子の発現を網羅的に解析するものです。
*3 MICOS複合体
MICOS複合体は、Mitochondrial Contact Site and Cristae Organizing System (ミトコンドリア接触部位およびクリステ形成システム)の略称です。この複合体は、図3に示すような複合体であり、ミトコンドリアの内膜に位置し、クリステと呼ばれるミトコンドリア内膜の折り畳み構造の形成と維持に不可欠です。
*4 AlphaFold
AlphaFoldは、GoogleのDeepMindによって開発された人工知能システムです。このプログラムは、タンパク質の折り畳み構造を原子レベルの精度で予測することができます。2024年のノーベル化学賞の受賞対象となりました。
<参考文献>
Kishita Y, Shimura M, Kohda M, Akita M, Imai-Okazaki A, Yatsuka Y, Nakajima Y, Ito T, Ohtake A, Murayama K, Okazaki Y. A novel homozygous variant in MICOS13/QIL1 causes hepato-encephalopathy with mitochondrial DNA depletion syndrome. Mol Genet Genomic Med. 2020;8(10):e1427. doi:10.1002/mgg3.1427
■ 原著論文
本研究はLiver International 誌のオンライン版で(2024年11月8日付)先行公開されました。
タイトル: Variants in MICOS10 identified by whole genome sequencing and RNA sequencing in a new type of hepatocerebral mitochondrial DNA depletion syndrome
タイトル(日本語訳): 肝-神経型ミトコンドリアDNA枯渇症の原因としてMICOS10 遺伝子のバリアントを全ゲノム解析とRNAシークエンスによって同定した
著者: Yoshihito Kishita1,2, Ayumu Sugiura2, Nanako Omichi1, Masaru Shimura3, Yukiko Yatsuka2, Kohta Nakamura2, Toju Tanaka4, Mitsuru Kubota5, Kei Murayama2,6, Akira Ohtake7, Yasushi Okazaki2,8
著者(日本語表記): 木下善仁1,2, 杉浦歩2, 大道納菜子1, 志村優3, 八塚由紀子2, 中村弘太2, 田中藤樹4, 窪田満5, 村山圭2,6 , 大竹明7, 岡﨑康司2,8
著者所属: 1 近畿大学理工学部生命科学科、2 順天堂大学大学院 難治性疾患診断・治療学講座/難病の診断と治療研究センター、3 千葉県こども病院 代謝科、4 北海道医療センター小児遺伝代謝センター、5 国立成育医療研究センター総合診療部、6 順天堂大学 小児科、7 埼玉医科大学 小児科学・ゲノム医療学、8 国立研究開発法人 理化学研究所 生命医科学研究センター 応用ゲノム解析技術研究チーム
DOI: 10.1111/liv.16148
本研究は、JSPS科研費 基盤研究B(JP19H03624)、基盤研究A(JP23H00424)、基盤研究C(JP23K07236)、AMED難治性疾患実用化研究事業(JP22ek0109468)、AMEDゲノム創薬基盤推進研究事業( JP22kk0305015 )、東京生化学研究会(現:中外創薬科学財団)助成金、の支援を受け、多施設との共同研究のもとに実施されました。
また、研究実施に当たって、順天堂大学の研究基盤センター形態解析イメージング研究室とゲノム・再生医療センターの協力のもと実施されました。
なお、本研究にご協力いただいた皆様には深謝いたします。
このプレスリリースには、メディア関係者向けの情報があります
メディアユーザー登録を行うと、企業担当者の連絡先や、イベント・記者会見の情報など様々な特記情報を閲覧できます。※内容はプレスリリースにより異なります。
すべての画像
