系統的に新規なβ-1,2-グルカナーゼ群の発見 ~酵素の構造解析・機能解析から解明された分子進化の手がかり~
【研究の要旨とポイント】
β-1,2-グルカンの分解酵素について、アミノ酸配列の網羅的な相同性検索を行い、機能未知で系統的に新規な4つのグループ(Group 1~4)を発見しました。これらのうち3つのグループ(Group 1~3)でβ-1,2-グルカンを分解する活性を発見しました。
これらグループの酵素の立体構造と予測構造の解析、反応機構の解析、推定される触媒残基の位置の比較から、これら3つのグループが系統的に新規な糖質加水分解酵素(GH)のファミリー(*1)であることが示され、新規GHファミリーGH192、GH193、GH194が創設されました。
本研究のさらなる発展により、新規酵素の探索や酵素反応メカニズムの解明が進展し、新たなオリゴ糖合成のための酵素機能改変などへの応用が期待されます。
【研究の概要】
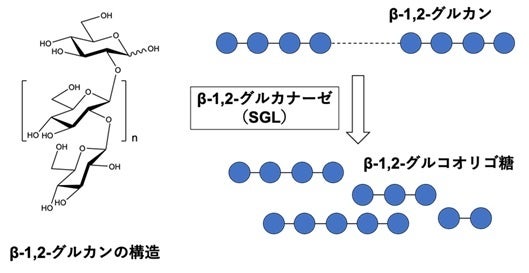
東京理科大学 創域理工学部 生命生物科学科の中島 将博准教授、同大学大学院 創域理工学研究科 生命生物科学専攻の元内 省博士(日本学術振興会特別研究員PD)、産業技術総合研究所 人工知能研究センターの小林 海渡研究員、新潟大学 農学部 農学科の中井 博之准教授の共同研究グループは、糖質加水分解酵素(Glycoside Hydrolase; GH)に関して、β-1,2-グルカンを分解してβ-1,2-グルコオリゴ糖を生成するGHファミリー(GH144、GH162)に属する酵素(β-1,2-グルカナーゼ, SGL)(図1)を基にしたアミノ酸配列の網羅的な相同性検索により、機能未知で系統的に新規な4つのグループ(Group1~4)を発見しました(図2)。機能と構造の側面から解析を行った結果、Group 1~3のタンパク質がSGLであることがわかりました。また、構造的な特徴や反応機構の比較から、Group 1~3は既知のGHファミリーであるGH144、GH162、GH189とは異なる新規ファミリーに分類されるべきであることが示され、このGroup 1~3に対してそれぞれGH192、GH193、GH194が新たに創設されました。
β-1,2-グルカンは細菌によって生産される天然のグルコースポリマーであり、共生因子、浸透圧調節、宿主免疫系の回避など、生理学的に重要な役割を果たしています。しかし、その希少さのため、β-1,2-グルカンの合成と分解に関与する酵素の研究例は限られており、関連酵素の自然界における分布の全体像の把握には至っておりません。本研究グループは、過去にさまざまなβ-1,2-グルカン関連酵素に関する研究を行い、酵素がはたらく分子メカニズムを解明してきました。今回、さらに理解を深めるべく、土壌細菌Chitinophaga pinensis由来のSGL(CpSGL)と、糸状菌Talaromyces funiculosus由来のSGL(TfSGL)を用いた相同性検索を行い、既知のGHファミリー周辺の探索を行いました。
その結果、系統的に全く新しい4つのグループ(Group 1~4)を発見しました。これらのタンパク質について詳細に解析したところ、Group 1~3のタンパク質はβ-1,2-グルカンを特異的に分解する作用を示しました。Group 4のタンパク質については分解対象となる基質を同定することができませんでした。Group 1~3の酵素について、反応速度論的解析や酵素特性解析を行ったところ、既知のGH144やGH162に属するSGLと類似した性質を示したことから、今回発見した酵素もSGLであると結論付けられました。さらに、これら3つのGHファミリーはいずれも既知のSGLと同様の全体構造[(α/α)6バレル構造]を有し、アノマー反転型(*2)の反応機構を示すことから、構造分類群としてはGH144やGH162と同じくclan GH-Sに属することを見出しました。しかし、推定される触媒残基位置の違いや系統樹における位置関係から、Group 1~3は既知のGHファミリーとは異なる新規ファミリーであることが示され、それぞれのグループ対してGH192、GH193、GH194という新しいGHファミリーが創設されました。
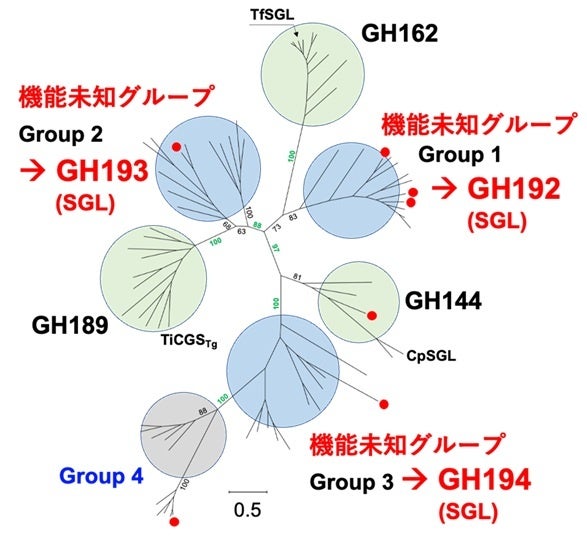
赤丸は本研究で使用したタンパク質を示している。それらの由来の菌はGroup 1、グラム陰性菌Photobacterium gaetbulicola、グラム陰性菌Endozoicomonas elysicola;Group 2、グラム陽性菌Sanguibacter keddieii;Group 3、P. gaetbulicola;Group 4、P. gaetbulicola;GH144、植物病原菌Xanthomonas campestris pv. campestris。
本研究成果は、2025年5月24日に国際学術誌「Protein Science」にオンライン掲載されました。
【研究の背景】
β-1,2-グルカンは細菌によって生産される天然のグルコースポリマーであり、共生因子、浸透圧調節、宿主免疫系の回避など生理的に重要な役割を果たしています。細菌における役割だけでなく、β-1,2-グルカンの分解産物であるβ-1,2-グルコオリゴ糖が植物において免疫を誘導することも報告されています。β-1,2-グルカンの合成酵素としては、日和見感染菌Brucella abortus、根粒菌Sinorhizobium meliloti、植物病原菌Rhizobium radiobacter由来の環状β-1,2-グルカン合成酵素(CGS)が報告されています。分解酵素としては、2014年のグラム陽性細菌Listeria innocua由来1,2-β-オリゴグルカンホスホリラーゼの遺伝子同定が初めての報告になります。その後、この酵素を利用したβ-1,2-グルカンの大量合成法が確立されました。このβ-1,2-グルカンを用いて、β-1,2-グルカンからβ-1,2-グルコオリゴ糖を遊離するエンド型酵素(*3)であるβ-1,2-グルカナーゼ(SGL)が、細菌および糸状菌それぞれから遺伝子同定されています。これらの酵素はGHファミリーに属しており、細菌由来SGLはGH144、糸状菌由来SGLはGH162として新規なGHファミリーが創設されました(※1)。近年では、好熱性細菌Thermoanaerobacter italicus由来のCGSの機能未知ドメイン(TiCGSTg)が糖転移反応により直鎖状糖鎖を環状化できることが明らかになり、このドメインのグループに対しても新たなGHファミリー(GH189)が創設されました(※2)。
このように、近年さまざまなβ-1,2-グルカン関連酵素が新たに発見され、研究が大きく進展してきました。特に、新規GHファミリーの発見や生化学的機能の解明が進み、β-1,2-グルカン代謝に関する理解が深まっています。しかし、β-1,2-グルカン代謝関連遺伝子が広く微生物に存在し、β-1,2-グルカンが植物を含む生物間の相互作用に関わっていることを踏まえると、これらの酵素の分布や多様性、生理的役割についての理解はまだ十分とは言えません。
そこで今回、GH144に属するChitinophaga pinensis由来のSGL(CpSGL)と、GH162に属する糸状菌Talaromyces funiculosus由来のSGL(TfSGL)に着目して、新たな分解酵素の検索を行いました。そして、発見した酵素の生化学的解析および構造解析を通じて、触媒残基の配置の変化や基質認識機構の相違など、反応機構の変化を伴う複雑な分子進化過程を解明することを本研究の主要な目的としました。
※1: 日本経済新聞プレスリリース(2019年3月30日付)
※2: 東京理科大学プレスリリース(2024年2月13日付)
『β-1,2-グルカンの環化に関わるメカニズムを解明 ~生理学的に重要な環状β-1,2-グルカン研究の進展に寄与~』
【研究結果の詳細】
①SGLのGHファミリーにおけるアミノ酸配列の相同性検索
CpSGL (GH144)とTfSGL (GH162)をそれぞれクエリとしてPSI-BLASTを用いた相同性検索を行いました。その結果、GH144、GH162、GH189は互いに遠縁の関係にあっただけでなく、これまで報告されていなかった4つの機能未知のGroup 1~4が見出されました。また、これらがGH144、GH162、GH189とともに、巨大な系統群(SGLクラン)を形成していることがわかりました(図2)。
SGLクランの各グループから代表的な酵素を選定して以降の解析に用いました。また、基質の結合のポーズについては、植物病原菌Xanthomonas campestris由来のGH144 SGL (XcSGL)とβ-1,2-グルカンとの複合体構造の取得を行い、この構造との比較により考察を行いました。
②機能未知グループの各酵素の機能探索
β-1,2-グルカンに対する加水分解活性は、Group 1~3の酵素で確認されました。これらの酵素は全てβ-1,2-グルカンに特異的でしたが、最適pHや糖鎖の分解パターンはそれぞれ異なることが明らかになりました。一方で、Group 4については反応を示す基質は見出されませんでした。Group 1~3の酵素については反応速度論的解析も行い、これらの酵素がGHファミリーの酵素として十分に高い加水分解速度を有することが示されました。これらの結果から、Group 1~3という機能未知グループの真の酵素機能を発見したと言えます。
GHファミリーの酵素は基本的に2種類の反応機構(アノマー保持型とアノマー反転型、*2)のいずれかを有しています。Group 1~3の酵素の反応機構における基礎的な性質を決定するため、これらの酵素によるβ-1,2-グルカンの加水分解過程における旋光度変化を解析しました。その結果、これら3つの酵素はいずれもTfSGLやCpSGLと同様の変化を示し、アノマー反転型で反応が進行することが明らかになりました。
③SGLクランの構造的特徴
Group 1~4の酵素の立体構造を詳細に分析し、いずれも既知のGH144, GH162, GH189と同様の(α/α)6バレル構造を有することを明らかにしました。新たに発見した酵素と既知GHファミリーの酵素との構造比較では、α-ヘリックスの位置や長さに特徴的な違いが見られ、特にGH189には固有の挿入配列があり、それが構造的特徴となっていることが見出されました。これらの構造的差異は、酵素の進化的起源や機能の多様化に関連している可能性があります。
また、SGLクラン全体で保存されている重要なアミノ酸残基として、(XcSGL (GH144)の残基番号で)E239、Y367が見出されました。F286もGH162以外の全てで保存されている残基でした。これらは全て基質ポケットに存在する残基であり、E239は触媒残基の一つ、Y367は基質と相互作用をする残基、F286は酵素反応の際に基質と反応する水分子または触媒残基の近傍にあり、全て十分な酵素機能の発揮に重要と考えられます(図3)。これらの残基に相当するアミノ酸は、SGLクランに属するタンパク質を特徴づける指標として利用できます。さらに、このような保存性は機能未知のGroup 4のタンパク質においても認められました。
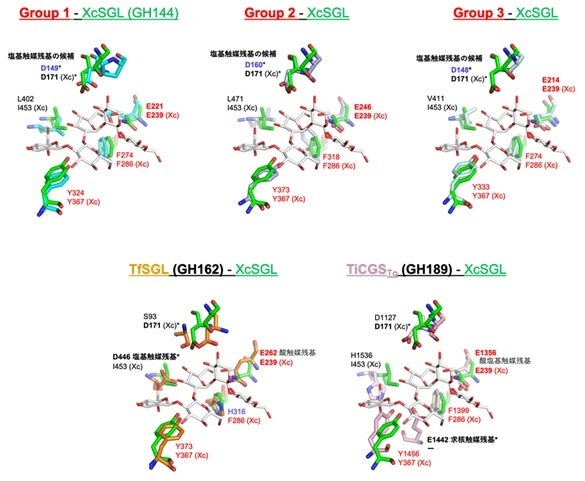
上段は各グループ、ファミリーの残基、下段はXcSGL(GH144)の残基を記載している。緑と白のスティック表示はそれぞれXcSGLの残基と基質の一部を示している。その他の色のスティック表示は各グループ、ファミリーの残基を示している。赤でラベルした残基はSGLクランを特徴付ける残基。触媒残基と触媒残基の候補は太字で示し、日本語でラベルしている。塩基触媒残基(GH189の場合は求核触媒残基)またはその候補の残基は、それらの位置の比較のためにアスタリスクを添えている。Group 1はE . elysicola由来SGLの構造である。右下E1442の下の – は対応する残基がないことを示している。
④触媒残基の位置とGHファミリーの分類
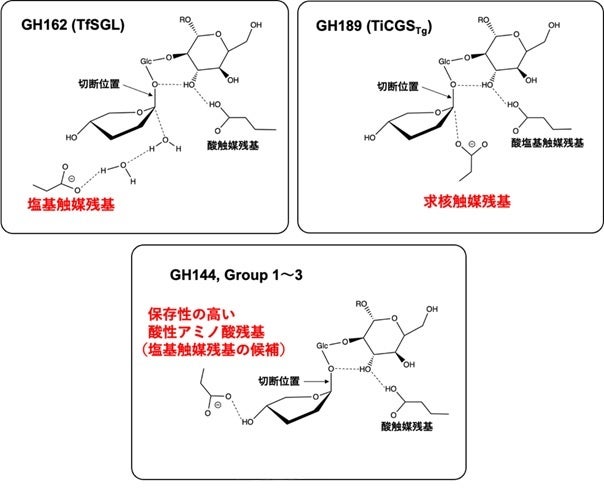
GH酵素のファミリー間における本質的な相違は、その一つとして触媒残基の位置に反映されるため、それらの位置は分子進化を知る上での有力な情報です。一般的にGH酵素は2個の酸性アミノ酸残基が触媒残基となります(アノマー反転型の場合は一般酸触媒残基と一般塩基触媒残基、以下は一般の記載を省略)。そこで、SGLクランにおいて機能が明らかになった各グループの酸性アミノ酸残基の位置を比較しました。TfSGL(GH162)で同定されている酸触媒残基(E262)はSGLクランの全グループで保存されていました(図3、4)。これは、先述のSGLクランに属するタンパク質を特徴づける指標となる残基の一つです。Group 1~3の酵素における対応する残基はその変異体で活性をほぼ失ったため、これらの残基は酸触媒残基であることが示唆されました。
一方で、塩基触媒残基については、一般的なパターンである求核水に直接相互作用する酸性アミノ酸残基が存在しませんでした。しかし、GH144酵素の基質ポケット中で保存されている酸性アミノ酸残基がGroup 1~3に保存されており(図3、4)、その変異体は活性をほぼ失いました。したがって、これらの保存残基は触媒残基の有力な候補であり、反応機構はGH144とGroup 1~3で共通しているものと推定されます。ただし、これらの残基の位置は特殊なため、触媒残基として確定するにはシミュレーションなどの他の証拠も必要です。GH162の塩基触媒残基やGH189の求核触媒残基(アノマー保持型における塩基触媒残基に対応する残基)は、Group 1~3では対応する位置に触媒可能な残基が存在していませんでした(図3、4)。これは、Group 1~3の塩基触媒残基の位置が確定しているかどうかにかかわらず、Group 1~3とGH162、GH189で反応機構が明らかに異なっていることを示しています。系統樹では反応機構ごとにグループが配置されてはおらず、Group 1とGroup 2はそれぞれGH162、GH189と近いところに位置しています。アミノ酸配列、立体構造、反応機構の情報を総合した結果、Group 1~3は既存のGHファミリーの拡張ではなく新規のGHファミリーと認定されました。
本研究では、これまで機能が不明だった4つのGroupのうち3つについて、生理学的機能と立体構造を解明し、Group 1~3それぞれに対して新たなGHファミリー(GH192、GH193、GH194)が創設されました(図2)。SGLクランではアミノ酸配列の同一性が極めて低く、保存残基も少ないことから、分子進化の過程で大きな構造的・機能的多様化が生じたと考えられます。このように、SGL関連酵素の広範な分布と多様性は、自然界におけるβ-1,2-グルカン代謝の重要性と進化的複雑性を示しています。
本研究を主導した中島准教授は、「糖鎖はさまざまな生理機能を持っていますが、その構造が複雑で調製が難しく、機能の解明が容易でないものが多くあります。それゆえに、そういった糖鎖に作用する新たな分解酵素の探索も同様に困難な課題の1つです。糖鎖を合成する際には酵素を用いた方法が広く採用されており、新たな分解酵素が発見されれば新たな糖鎖の合成に応用できる可能性を秘めています。このような背景から、新しい分解酵素の探索研究は非常に重要だと考えています。系統的に新たな酵素群が見つかれば、それを起点にさらなる新規酵素の発見につながるため、本研究成果には大きな意義があると考えています」と、コメントしています。
※本研究は、日本学術振興会(JSPS)の科研費(23K05041)、科学技術振興機構(JST)の次世代研究者挑戦的研究プログラム(SPRING, JPMJSP2151)の助成を受けて実施したものです。
【用語】
※PR TIMESの文字数制限により割愛しております。用語詳細は東京理科大学WEBページ(https://www.tus.ac.jp/today/archive/20250602_9384.html)をご参照ください。
【論文情報】
雑誌名:Protein Science
論文タイトル:New glycoside hydrolase families of β-1,2-glucanases
著者:Masahiro Nakajima, Nobukiyo Tanaka, Sei Motouchi, Kaito Kobayashi, Hisaka Shimizu, Koichi Abe, Naoya Hosoyamada, Naoya Abara, Naoko Morimoto, Narumi Hiramoto, Ryosuke Nakata, Akira Takashima, Marie Hosoki, Soichiro Suzuki, Kako Shikano, Takahiro Fujimaru, Shiho Imagawa, Yukiya Kawadai, Ziyu Wang, Yoshinao Kitano, Takanori Nihira, Hiroyuki Nakai, Hayao Taguchi
【発表者】
中島 将博 東京理科大学 創域理工学部 生命生物科学科 准教授 <責任著者>
元内 省 東京理科大学 創域理工学部 生命生物科学科 日本学術振興会特別研究員PD
小林 海渡 産業技術総合研究所 人工知能研究センター 研究員
中井 博之 新潟大学 農学部 農学科 准教授
【研究に関する問い合わせ先】
東京理科大学 創域理工学部 生命生物科学科 准教授
中島 将博
E-mail: m-nakajima【@】rs.tus.ac.jp
【報道・広報に関する問い合わせ先】
東京理科大学 経営企画部 広報課
TEL: 03-5228-8107 FAX: 03-3260-5823
E-mail: koho【@】admin.tus.ac.jp
産業技術総合研究所 報道室
TEL: 029-862-6216 FAX: 029-844-8028
E-mail: hodo-ml【@】aist.go.jp
新潟大学 広報事務室
TEL: 025-262-7000
Email: pr-office【@】adm.niigata-u.ac.jp
【@】は@にご変更ください。
※PR TIMESのシステムでは上付き・下付き文字や特殊文字等を使用できないため、正式な表記と異なる場合がございますのでご留意ください。正式な表記は、東京理科大学WEBページ(https://www.tus.ac.jp/today/archive/20250602_9384.html)をご参照ください。
すべての画像
